Программа «МИКРОБ-АВТОМАТ» для автоматизации проведения микробиологических исследований
Внедрение ридеров для выполнения ежедневных самых трудоемких и дорогостоящих исследований по идентификации и определению антибиотикочувствительности в работу микробиологических лабораторий несомненно позволяет получать стандартные результаты, ускоряет выдачу анализов, вытесняет малопроизводительный ручной труд.
Мы отдаем предпочтение ридерам фирмы THERMO-Electron (Финляндия), так как они легко адаптируются как к отечественным, так и к импортным (например, производства фирмы PLIVA-Lachema, Чехия) тест-системам для проведения идентификации бактерий и определения антибиотикочувствительности с набором антибиотиков по желанию заказчика. Возможность использования тест-систем не только фирм-производителей ридера избавляет от безвыходных ситуаций, оставляет возможность выбора тест-систем исходя из их качества, цены, доступности для каждого пользователя.
На базе приборов iEMS-Reader и Multiskan-Ascent нами создано «Автоматизированное рабочее место микробиолога, химиотерапевта «МИКРОБ-АВТОМАТ». Созданная для этих целей программа «МИКРОБ-АВТОМАТ» позволяет проводить автоматизированное считывание результатов биохимических тестов в вышеуказанных микротест-системах. Все дополнительные тесты рекомендуется вносить вручную, как описано в предыдущей статье.
а) Идентификация микроорганизмов различных групп. Компьютерные банки данных и алгоритм обработки в программе идентичны таковым в программе «Идентификация».
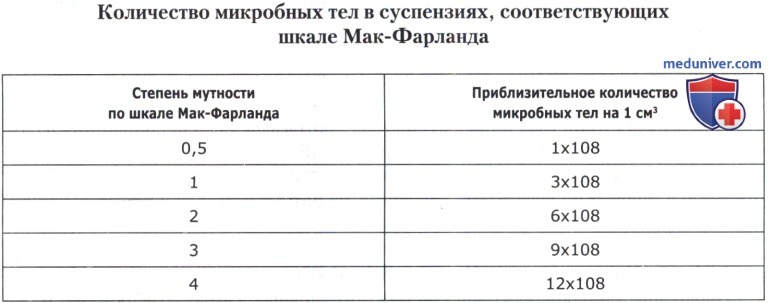
При автоматизированном считывании результатов биохимических реакций в коммерческих тест-системах предъявляются более жесткие требования к приготовлению суспензий, калибровку которых желательно проводить на том же ридере, на котором проводится считывание тест-систем. В инструкциях к микроба-тестам фирмы PLIVA-Lachema, Чехия, указывается, что мутность исходной суспензии должна соответствовать степени мутности по шкале Мак-Фарланда. В таблице ниже приведено приблизительное количество микробных тел на 1 см3 в суспензиях, соответствующих степеням мутности по шкале Мак-Фарланда.
Например, в инструкции к STREPTOtestl6 сказано, что мутность суспензии исследуемого штамма должна соответствовать третьей степени мутности по шкале Мак-Фарланда или содержать 9х108 микробных тел на 1 см3. Для вычисления количества микробных тел в единице объема в программе «МИКРОБ-АВТОМАТ» используется специальная калибровочная зависимость. Эта функция основана на экспериментально полученной зависимости оптической плотности суспензии микроорганизмов при длине волны фотометрирования 620 нм от количества микробных тел в 1 см3 суспензии.
б) Методика выполнения теста «Калибровка суспензий». Готовят суспензии исследуемых культур и при помощи пипеточного дозатора в количестве 100 мкл вносят в ячейки планшета. Для определения мутности приготовленных суспензий необходимо загрузить тест для измерения суспензий через команду основного меню «Загрузить тест ...».
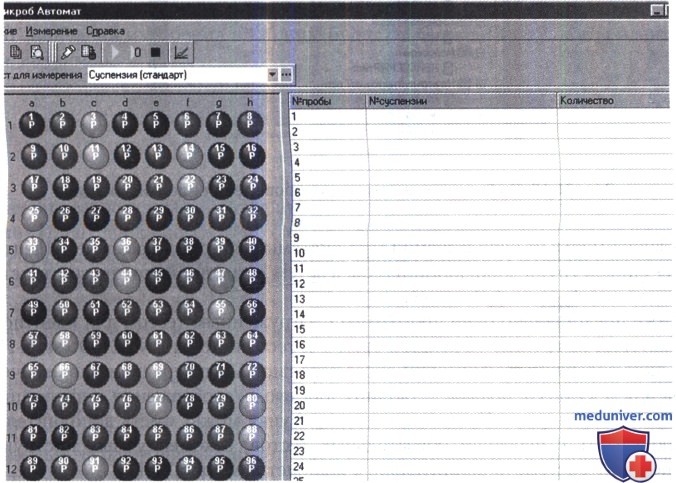
Ниже представлено окно программы с загруженным тестом для измерения суспензий. Левая часть окна содержит графический образ конфигурации теста для измерений в зеркальном отображении, правая — таблицу для ввода/отображения информации при измерении суспензий.
В одном планшете можно провести калибровку 96 суспензий.
После проведения измерения программа выдает результаты в следующем виде, они автоматически записываются в архив измерений.
Для идентификации микроорганизмов загружается соответствующий тест через команду основного меню «Загрузить тест...».
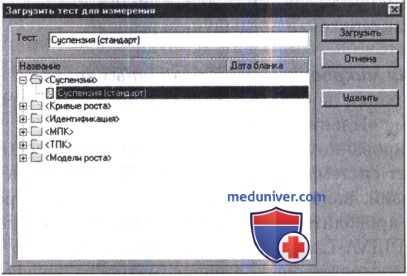
В зависимости от используемой тест-системы выбирают в разделе «Идентификация» список групп микроорганизмов, а затем в заданной группе соответствующую тест-систему и загружают ее.
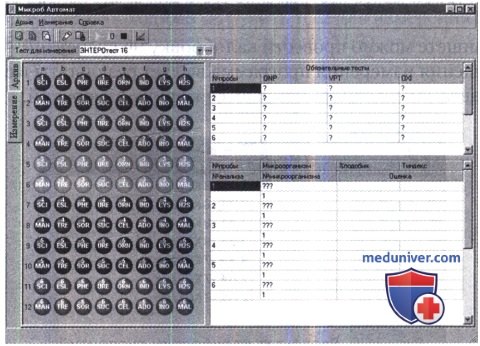
Ниже представлено окно программы с загруженным тестом для идентификации бактерий из группы «Оксидаза-Грам-» и тест-системы ENTEROtestl6. Левая часть окна содержит графический образ тест-системы с перечнем биохимических тестов для 6 культур, правая — таблицу для ввода/отображения информации об испытуемых культурах. Правая половина рисунка может иметь дополнительную верхнюю часть с перечнем обязательных тестов, рекомендуемых инструкцией к данной тест-системе дополнительно к планшетным.
Для идентификации микроорганизмов заполняются поля в колонке «№ анализа» и «№ микроорганизма». Если при идентификации культур проводились дополнительные обязательные тесты, их результаты вручную вводятся в верхнее поле таблицы соответственно номеру пробы и наименованию теста. При вводе значений этих тестов используются те же знаки: [+], [-] или [?], значения которых приведены в предыдущем разделе.
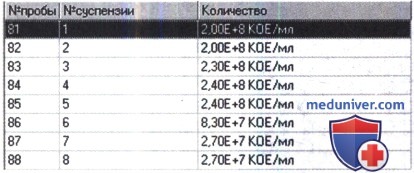

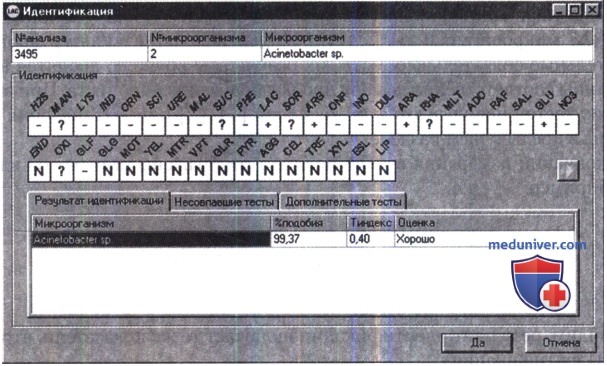
По окончании измерения в таблице будут отображены результаты идентификации, например:

Как видим, результат идентификации включает те же показатели, что и в программе «Идентификация».
Одновременно с выводом полученных данных в таблицу в графическом образе теста отображаются результаты измерения — значения [+] или [-] отображаются в правом верхнем углу изображения лунки для каждого теста:

При невозможности программой идентифицировать микроорганизм на базе полученных результатов в поле микроорганизма вместо его наименования будет выдано сообщение «Не идентифицирован».
Результаты идентификации автоматически сохраняются в архивах измерений и анализов. Это предусмотрено на случай возможной коррекции результатов нечетких тестов при визуальной оценке, внесения результатов дополнительных тестов, рекомендованных программой, с целью получения повторного расчета результата идентификации с использованием более широкого спектра признаков, а также сохранения данных для последующего анализа. В том случае, когда после измерения тест-систем для идентификации микроорганизмов программа выдает сообщение «Не идентифицирован» или пользователь хочет уточнить или просто просмотреть результаты идентификации, вызывается информация о выполненном анализе из архива измерений и архива анализов.
На экранной форме соответственно номеру вызванного анализа появляется полученный биохимический профиль по результатам планшетных и дополнительных обязательных тестов, если таковые выполнялись.
Результат идентификации — название микроорганизма — появляется сверху в поле «Идентифицированный микроорганизм». В случае невозможности идентификации по результатам выполненных тестов в данном поле появляется надпись «Не идентифицирован». При этом в таблице расчетных данных под приведенным профилем может быть два варианта информации: в первом варианте — список возможных культур пуст (это возможно при работе со смешанной культурой или если такой культуры нет в банке данных); во втором варианте — несколько культур с параметрами идентификации (% подобия и Т-индекс) ниже уровня, заложенного в программе. В этом случае по каждому микроорганизму программа в результате обработки введенной информации выдает следующие данные: «Результат идентификации», «Несовпавшие тесты» и «Дополнительные тесты». При этом результат идентификации выведен на экран, а просмотр данных по несовпавшим (атипичным) и дополнительным тестам обеспечивается нажатием на одноименные закладки. После уточнения идентификации (повторный расчет идентификации после введения выполненных рекомендованных дополнительных тестов и коррекции нечетких тестов) полученный результат автоматически запишется в архив анализов. Экспорт данных в «Систему микробиологического мониторинга «МИКРОБ-2» из архива анализов автоматизирован.
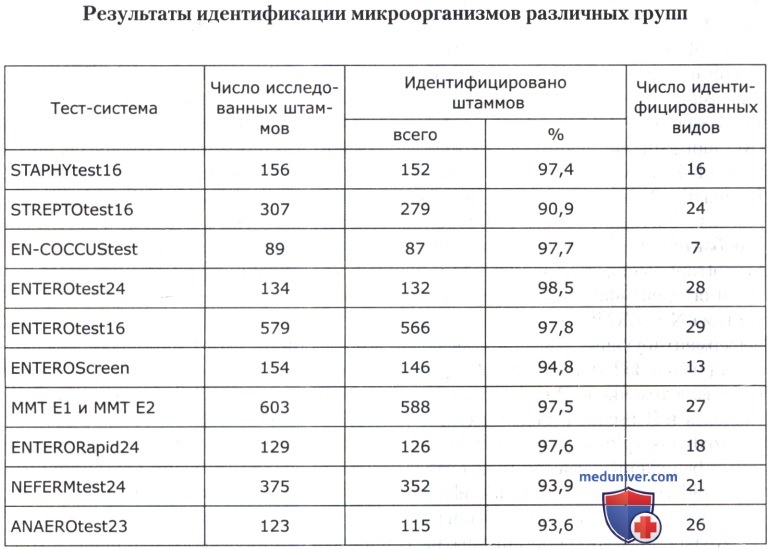
В таблице ниже приведены обобщенные результаты идентификации 2860 клинических штаммов бактерий из различных групп, биохимическая активность которых исследована с использованием коммерческих тест-систем, а обработка проведена при помощи программы «Идентификация» при визуальном считывании и программы «МИКРОБ-АВТОМАТ» при автоматизированном считывании результатов биохимических реакций.
Из приведенных данных видно, что в зависимости от использованных тест-систем процент идентификации варьировал от 90,9 до 98,5 %.
Оценка отдельных тест-систем показала, что используя STAPHYtest16, предназначенного для рутинной идентификации стафилококков и дифференциации их с другими родственными грамположительными кокками, идентифицировано 152 (97,4 %) из 156 исследованных штаммов. В рамках данного исследования было идентифицировано 17 видов различных грамположительных кокков, из них 15 видов стафилококков: S. epidermidis (65 штаммов), S. aureus (21), S. haemolyticus (18), S. warneri (17), S. hominis (13), S. saprophyticus (2), S. auricularis (2), S. cohnii/ cochnii и S. cohnii/ureolyticus (5), S. capitis и S. capitis/ureolyticus (4) и др.
Для идентификации каталазоотрицательных грамположительных кокков в настоящее время фирма PLIVA-Lachema (Чехия) выпускает отдельные тест-системы: STREPTOtestl6 для идентификации наиболее часто вызывающих заболевания у человека представителей микроорганизмов из группы пиогенных, оральных и других стрептококков, и EN-COCCU Stest для идентификации энтерококков. В качестве разделительного теста при выборе тест-системы предложен полосочный PYRtest для быстрого (5-10 мин) выявления цветной реакции. Данный тест дает положительную реакцию на абсолютное большинство клинически значимых энтерококков, из стрептококков — только на S. pyogenes.
Оценка STREPTOtestl6 проведена по результатам исследования 307 культур, выделенных из клинического материала, из которых идентифицировано 279 штаммов (90,9 %), относящихся к 24 видам различных стрептококков: S. pyogenes (28 штаммов), S. pneumoniae (23), S. oralis/mitis 1 и S. mitis 2 (51), S. agalactiae (22), S. anginosus (11), S. equi/equi (15), S. uberis (5), S. parauberis (9), S. salivarius (25), S. sanguis (13), S. mutans gr. (6), S. dysgalactiae/equisimilis и S. dysgalactiae/desgalactiae (21), S. equinus (9), S. bovis (7) и др.
С использованием EN-COCCUStest из 89 клинических штаммов идентифицировано 87 культур (97,7 %), включающих 7 видов: Е. faecalis (55 штаммов), Е. faecalis assaccharolyticus (4), Е. faecium (15), Е. durans (8), Е. avium (3), Е. gallinarum и Е. raffinosus (по 1 штамму).
Для идентификации грамотрицательных ферментирующих бактерий наряду с ENTEROtestl6 в настоящее время предложены модифицированная тест-система ENTEROtest24 и две новые тест-системы для ускоренной (за 4 часа) идентификации энтеробактерий ENTERORapid24 и в основном для уропатогенных культур ENTEROScreen. При исследовании 134 клинических штаммов при помощи ENTEROtest24 идентифицировано 132 культуры (98,5 %), включающие 28 видов различных энтеробактерий и оксидазоотрицательных неферментирующих бактерий. Несколько меньший уровень идентификации примерно с тем же спектром культур получен при использовании тест-системы ENTEROtestl6 (исследовано 579 штаммов) и отечественных тест-систем ММТ Е1 и ММТ Е2 (исследовано 603 штамма) — идентификация в пределах 97,8 % и 97,5 % соответственно. Аналогичные результаты выявлены при использовании тест-системы ENTERORapid24 (97,6 %).
Тест-система ENTEROScreen была использована при исследовании 154 штаммов энтеробактерий, выделенных из мочи; проидентифицировано 146 культур (94,8 %), представленных 13 видами. В целом спектр культур, идентифицированных при помощи этих тест-систем, включал представителей 15 родов бактерий, в том числе Citrobacter (С. freundii — 22 штамма, С. koseri — 3, С. amalonaticus, С. braakii, С. youngae, С. werkmanii — по 1 штамму), Enterobacter (Е. cloacae—106, Е. aerogenes —23, Е. intermedius — 2 штамма), Escherichia (Е. coli — 437, Е. coli inactivae — 24, Е. hermannii, Е. fergusonii — по 1 штамму), Hafnia (Н. alvei — 3 штамма), Klebsiella (К. pneumoniae/pneumoniae —294, К. pneumoniae/ozaenae — 11, К. oxytoca —86 штаммов), Leclercia (L. adecarboxilata — 2 штамма), Morganella (М. morganii/morganii—19, М. morganii/sibonii — 4 штамма), Proteus (Р. mirabilis—147, Р. vulgaris —56, Р. Penneri — 6 штаммов), Providencia (Р. rettgeri —3, Р. stuartii —2 штамма), Salmonella— 12штаммов, Shigella— 1 штамм, Serratia(S. marcescens — 35, S. liquefaciens — 5, S. rubidaea, S. plymuthica no 2 штамма) и другие энтеробактерии, а также из неферментирующих бактерий — Acinetobacter sp. — 113, Stenotrophomonas maltophilia— Ии Flavimonas oryzihabitans — 3 штамма.
Для идентификации неферментирующих грамотрицательных бактерий тест-система NEFERMtest24 использована при исследовании 375 штаммов различных неферментирующих бактерий, выделенных из клинического материала. Идентифицировано 352 культуры (93,9 %), относящиеся к 21 виду микроорганизмов этой группы, в том числе Pseudomonas aeruginosa (172 штамма), Р. fluorescens (7), Р. alcaligenes и Р. pseudoalcaligenes (9), Stenotrophomonas maltophilia (39), Alcaligenes xylosoxidans/xylosoxidans и A. xylosoxidans/denitrificans (34), A. faecalis (3), Acinetobacter baumannii и calcoaceticus (33), A. haemolyticus (8), A. lwoffii (4), A. junii (2), Burkholderia cepacia (12), Bordetella bronchiseptica (4), Moraxella nonliquefaciens (4), Chryseobacterium meningosepticum (2), Ralstonia pickettii (2) и др.
Анаэробные микроорганизмы, выделенные из клинического материала в Институте хирургии им. А. А. Вишневского и в ЦИТО, идентифицировали при помощи ANAEROtest23. При этом из 123 штаммов верифицировано 115 (93,5 %), в том числе 28 штаммов из группы анаэробных кокков (7 видов), 24 — грамположительных неспорообразующих палочек (5 видов) и 5 — спорообразующих палочек (3 вида), 58 — грамотрицательных палочек (11 видов).
Все изученные тест-системы сертифицированы Министерством здравоохранения и социального развития РФ для автоматизированного считывания при помощи планшетных фотометров iEMS-Reader и Multiskan-assent, а также визуального считывания реакций биохимических тестов.
- Читать далее "Определение антибиотикочувствительности с использованием МПК-тестов"
Редактор: Искандер Милевски. Дата публикации: 4.09.2019
- Идентификация микроорганизмов с использованием коммерческих микротест-систем
- Автоматизация и компьютеризация при проведении микробиологических исследований
- Программа «Идентификация» для автоматизации проведения микробиологических исследований
- Программа «МИКРОБ-АВТОМАТ» для автоматизации проведения микробиологических исследований
- Определение антибиотикочувствительности с использованием МПК-тестов
- Определение антибиотикочувствительности с использованием ТПК-тестов
- Решение практических задач в клинической микробиологии и химиотерапии
